2024年2月,醫學部卡爾森國際腫瘤中心朱衛國特聘教授團隊在《Cell Reports》雜志上發表題為“The ARID1A-METTL3-m6A axis ensures effective RNase H1-mediated resolution of R-loops and genome stability”的研究論文,揭示了R-loop中RNA發生的m6A甲基化修飾對于RNase H識別R-loop并驅動R-Loop清除起到十分關鍵的作用。醫學部助理教授張俊,研究生陳鳳為本研究共同第一作者,朱衛國特聘教授為通訊作者。

R-Loop是新轉錄生成的RNA鏈與DNA模板鏈形成的三鏈結構,這種結構在生物學中具有重要的功能,但R-Loop在特定基因組位點的過度累積會引發基因組的不穩定性。細胞內存在多種機制防范R-Loop的過度積累,其中RNaseH被公認為降解R-Loop的關鍵分子。然而,目前人們尚不清楚R-Loop是如何被RNase H精確地識別和分解,并最終達到維持基因組穩定性的目的。
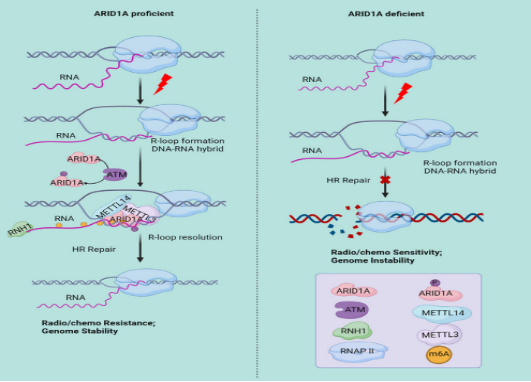
本研究首先證實了RNase H1RNase H1更傾向結合帶有m6A甲基化修飾的R-Loop,并推動R-Loop的分解以促進DSB的DNA末端切除,從而確保基因組的穩定性。機制上,該團隊鑒定出ARID1A在響應DNA損傷應激時快速識別R-Loop并進一步招募RNA甲基轉移酶METTL3/14,從而驅動R-LoopRNA發生m6A修飾。功能研究顯示ARID1A及METTL3的缺失會導致R-Loop積累,同時ARID1A-METTL3信號軸缺陷的細胞對基因毒性療法更加敏感。鑒于異常表達的ARID1A與腫瘤患者對基因毒性療法的耐藥性有關,這些發現為探索針對ARID1A異常表達癌癥的治療提供了潛在新策略(具體工作模式圖如下)。
本研究得到國家自然科學基金,廣東省自然科學基金以及深圳市醫學研究基金等項目的資助。
原文鏈接:https://www.sciencedirect.com/science/article/pii/S2211124724001074
(來源 深圳大學醫學部)





